Die Grundbausteine des Lebens
500ste Protein-Struktur mit Hilfe der Kristallografie an BESSY II entschlüsselt
Haare, Haut, Blutkreislauf und Nervensystem – alles wird von winzigen biologischen Bausteinen gesteuert. Wer nach diesen kleinsten Bausteinen des Lebens forscht, landet automatisch bei ihnen: den Proteinen. Um diese Grundbausteine des Lebens und die Vorgänge, die sie bewirken, besser zu verstehen, archivieren Wissenschaftler in aller Welt ihre Erkenntnisse über Proteine in einer gemeinsamen Datenbank. Die HZB-Forschungsgruppe Makromolekulare Kristallografie (MX) stellt Nutzern am BESSY II die Technik zur Verfügung, mit der sie Protein-Strukturen entschlüsseln können. Vor kurzem wurde die bereits 500ste Struktur an diesen MX-Strahlrohren des HZB von Wissenschaftlern der Bayer Healthcare Pharmaceuticals Berlin entschlüsselt.
Wer bei „Protein“ an jenen Stoff denkt, der Muskeln wachsen lässt, liegt nicht falsch, unterschätzt aber, wie vielfältig Proteine sind. Jeder Mensch hat rund 200.000 verschiedene im Körper und ohne sie läuft nichts. Daher gab ihnen ihr Entdecker Jöns Berzelius 1838 den lateinischen Namen „proteios“: erstrangig. Proteine erfüllen alle lebenswichtigen biologischen und chemischen Funktionen im Körper. Sie transportieren Sauerstoff im Blut, aktivieren als Botenstoffe Prozesse im Nervensystem, sind als Antikörper wichtiger Teil des Immunsystems und wachsen uns sogar aus dem Kopf – mal länger, mal kürzer. Elastische Spinnennetze, toxisches Schlangensekret, wasserabweisende Haut und leuchtende Glühwürmchen, all dies ermöglichen Proteine.
Proteine sind sehr große Moleküle. Sie setzen sich aus mindestens hundert Aminosäuren zusammen, häufig bilden sogar mehrere hundert Aminosäuren ein dreidimensionales Proteinmolekül. Die Reihenfolge der Aminosäuren, ist für die meisten Proteine bereits bekannt – für Wissenschaftler aber wenig aufschlussreich. Denn ob ein Protein beispielsweise als Hormon oder Enzym wirkt, ob es eine Reaktion katalysiert oder einen Stoff transportiert, wird nicht davon bestimmt, wie die Aminosäuren aneinander gereiht, sondern wie sie räumlich angeordnet sind. Die räumliche Struktur bestimmt also die Funktion, kann aber nicht aus der Sequenz abgeleitet werden.
Wissenschaftler nennen diese räumliche Anordnung die „Faltung“ des Proteins. Um daraus Rückschlüsse auf die Funktion des Proteins zu ziehen, untersuchen Forscher die Proteine auf atomarer Ebene. Dabei sind Skalpell und Mikroskop nutzlos. Nur mit intensivem Röntgenlicht wie aus der Synchrotronstrahlungsquelle BESSY II können Wissenschaftler die Moleküle von Atom zu Atom vermessen. Diese Methode nennt man Kristallografie. Hierfür züchten die Forscher aus den Proteinmolekülen zunächst Kristalle. Sie bestehen aus Billionen von identischen Proteinmolekülen, sind in sich symmetrisch und eignen sich daher besonders gut für die Untersuchung. Die Wissenschaftler beschießen diese Kristalle mit dem BESSY II-Synchrotronstrahl und erhalten ein sogenanntes Streubild. Es zeigt eine Vielzahl von Punkten aus denen sie, mit Hilfe einer mathematischen Operation, die Protein-Struktur rekonstruieren können.
Mit dieser Methode bestimmten die Forscher gemeinsam mit Wissenschaftlern der FU Berlin im Januar 2003 die erste Protein-Struktur an den Strahlrohren von BESSY II. Von da an ging es stetig aufwärts. Jahr für Jahr wurden mehr Strukturen an den Strahlrohren bestimmt, allein im Jahr 2010 weit über 100 Strukturen. Kürzlich gelang es Nutzern von Bayer Healthcare Pharmaceuticals Berlin die 500er Marke zu brechen. Die 500ste am BESSY II entschlüsselte Struktur gehört zum Protein PIM-1.
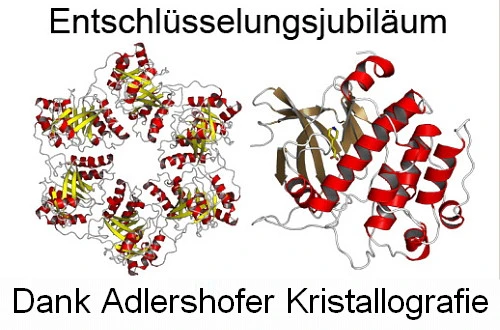
Struktur Nr. 500: Protein Kinase Pim-1
Pim-1 ist die 500ste kristalline Protein-Struktur, die an den BESSY II-Strahlrohren mit Hilfe der Kristallografie entschlüsselt wurde. Der Proteinkristall stammt aus dem Forschungslabor von Dr. Roman Hillig von Bayer Healthcare Pharmaceuticals in Berlin. Der untersuchte Kristall bestand aus dem Enzym Pim-1, das zusammen mit einem sogenannten Hemmstoff kokristallisiert wurde. Das Protein Pim-1 wird bei verschiedenen Krebsarten, wie zum Beispiel Prostata-Krebs und Lymphomen verstärkt produziert. Man vermutet deshalb, dass es ursächlich mit der Erkrankung in Zusammenhang steht und damit einen potentiellen Angriffspunkt für die Krebstherapie darstellt. Die am BESSY II entschlüsselte dreidimensionale Proteinstruktur könnte ein Ausgangspunkt für die Herstellung optimierter Pim-1-Hemmstoffe darstellen, aus denen schließlich ein Medikament für die Krebstherapie entwickelt werden soll. Diese müssen genau auf die Struktur des Proteins abgestimmt sein, um an diese „anzudocken“ und sie in ihrer Funktion zu blockieren.
1. Protein-Struktur die am HZB bestimmt wurde (2003):
Die erste Struktur, die an den Beamlines von BESSY II bestimmt wurde, war eine DNA Helikase (PDB Code 1NLF) und wurde im Januar 2003 in der internationalen „Protein Data Bank“ deponiert. DNA-Helikasen sind Enzyme, die für verschiedene Veränderungen an der menschlichen Erbinformation (DNA) verantwortlich sind. Sie sind beispielsweise für die Vervielfältigung der DNA unentbehrlich. Da es sich in diesem Fall um eine bakterielle Helikase handelte, hatte diese Arbeit vielleicht auch einen weitreichenden medizinischen Aspekt. Die Struktur wurde gemeinsam mit Norbert Sträter (heute Professor an der Uni Leipzig) und der Arbeitsgruppe von Wolfram Saenger (FU Berlin) gelöst.
1. Protein-Struktur, die überhaupt bestimmt wurde (1958):
Der britische Molekularbiologe John Kendrew klärte 1958 die erste dreidimensionale Struktur eines Proteins mit Hilfe der Röntgenkristallografie auf. Es handelte sich um Myoglobin, welches Sauerstoff innerhalb einer Zelle transportiert. Kendrew erhielt 1962 gemeinsam mit Max Perutz für die Aufklärung der dreidimensionalen Struktur von Myoglobin und Hämoglobin den Chemie-Nobelpreis.
Weitere Informationen:
Dr. Manfred Weiss
Tel.: (030) 8062-13149
E-Mail: manfred.weiss(at)helmholtz-berlin.de
Dr. Uwe Müller
Tel.: (030) 8062-14974
Fax: (030) 8062-14975
E-Mail: umue(at)helmholtz-berlin.de
Pressestelle:
Dr. Ina Helms
Tel.: (030) 8062-42034
Fax: (030) 8062-42998
E-Mail: ina.helms(at)helmholtz-berlin.de