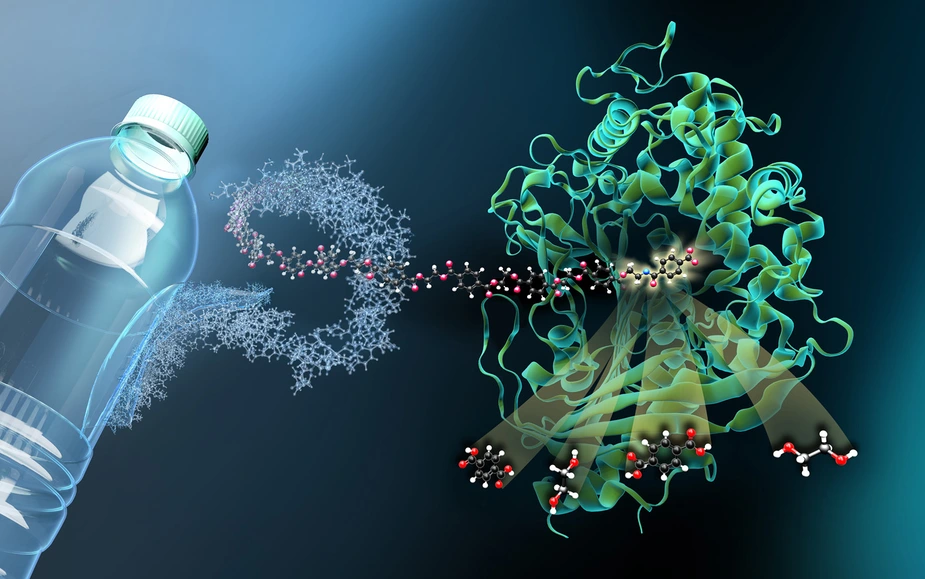
„Molekulare Schere“ für den Plastikmüll
Enzym zum Zerlegen von PET-Kunststoff an BESSY II entschlüsselt
Ein Team der Universität Greifswald und des Helmholtz-Zentrums Berlin (HZB) hat an BESSY II die Struktur eines wichtigen Enzyms ("MHETase") entschlüsselt. Die MHETase wurde in einem Bakterium entdeckt und ist in der Lage, zusammen mit einem zweiten Enzym, der PETase, den weit verbreiteten Kunststoff PET in seine Grundbausteine zu zerlegen. Die 3D-Struktur der MHETase ermöglichte es den Forschern bereits, die Aktivität dieses Enzyms gezielt zu optimieren, um es zusammen mit der PETase für das nachhaltige Recycling von PET zu nutzen. Die Ergebnisse wurden in der Fachzeitschrift Nature Communications veröffentlicht.
Kunststoffe sind wunderbare Materialien: extrem vielseitig und nahezu ewig haltbar. Doch genau das ist auch ein Problem, denn nach nur rund 100 Jahren Kunststoffproduktion befinden sich inzwischen Plastik-Partikel überall, im Grundwasser, in den Ozeanen, in der Luft und in der Nahrungskette. Nur ein winziger Bruchteil der Kunststoffe wird aktuell durch energie- und kostenintensive Verfahren recycelt, wobei sie bisher deutlich an Qualität verlieren oder wiederum von ‚frischem‘ Rohöl abhängen. Ein wichtiger industrieller Kunststoff ist PET, von dem jährlich ca. 50 Millionen Tonnen neu hergestellt werden.
2016 entdeckt: Bakterien auf PET
Erst 2016 hat eine Gruppe japanischer Forscher ein Bakterium entdeckt, das auf PET-Kunststoffen wachsen und sich teilweise davon ernähren kann. Wie sie herausfanden, besitzt dieses Bakterium zwei besondere Enzyme, die in der Lage sind, PET-Kunststoff abzubauen: PETase und MHETase. Zunächst zerlegt die PETase den Kunststoff in kleinere Bausteine, vorwiegend MHET, dann spaltet die MHETase diese in die zwei PET-Grundbausteine, Terephthalsäure und Ethylenglykol. Beide Bausteine sind sehr wertvoll für eine Neusynthese von PET – so dass erstmals ein nachhaltiger geschlossener Recyclingkreislauf – ohne Verwendung von Erdöl – möglich wird.
2018 PETase gelöst
Im April 2018 gelang es mehreren Arbeitsgruppen schließlich, die Struktur des Enzyms PETase aufzuklären, auch Experimente an der Lichtquelle Diamond waren daran beteiligt. Doch die PETase ist nur ein Teil der Lösung, genauso wichtig ist es, die Struktur des zweiten Enzyms, der MHETase, zu erhalten.
Jetzt: MHETase entschlüsselt
„Die MHETase ist deutlich größer als die PETase und noch komplexer. Ein einziges MHETase-Molekül besteht aus 600 Aminosäuren, das sind mehr als 4000 Atome. Die MHETase besitzt eine Oberfläche, die etwa doppelt so groß ist wie die von PETase. Damit gibt es auch wesentlich mehr Optionen, die Oberfläche dieses Enzyms zu modifizieren und für die Zerlegung von PET zu optimieren“, erklärt der Biochemiker und Strukturbiologe Dr. Gert Weber von der gemeinsamen Forschungsgruppe Proteinkristallographie am Helmholtz-Zentrum Berlin und der Freien Universität Berlin.
Als Vertretungsprofessor an der Uni Greifswald nahm Weber 2016 dort Kontakt mit Prof. Dr. Uwe Bornscheuer am Institut für Biochemie auf, der sich bereits mit plastikabbauenden Enzymen beschäftigte. Gemeinsam entwickelten sie die Idee, auch die Struktur der MHETase zu entschlüsseln und mit Hilfe dieser Einsicht das Enzym für den PET-Abbau zu optimieren. Dazu mussten sie zunächst das Enzym aus Bakterienzellen gewinnen und im Anschluss reinigen. In dieser Kooperation ist es nun gelungen, an der Berliner Synchrotronquelle BESSY II die dreidimensionale komplex gefaltete Architektur von MHETase zu entschlüsseln.
MHETase in Aktion
„Damit man in der Struktur sieht, wie das Enzym an PET bindet und es zersetzt, benötigt man ein Plastikfragment, das an MHETase bindet, aber nicht gespalten wird“, erklärt Weber. Ein Mitarbeiter aus dem damaligen Arbeitskreis Weber an der Universität Greifswald, Dr. Gottfried Palm, zerschnitt dafür eine PET-Flasche, zersetzte das PET chemisch und synthetisierte daraus ein kleines Plastikfragment, das an MHETase binden, aber von dieser nicht mehr gespalten werden kann. Aus dieser ‚blockierten‘ MHETase wurden dann winzige Kristalle für die Strukturuntersuchungen am HZB gezüchtet. „Durch diese Strukturuntersuchungen können wir der MHETase gewissermaßen ‚bei der Arbeit zuschauen‘ und daraus Strategien entwickeln, das Enzym zu optimieren“, erläutert Weber.
Besonderheiten identifiziert
„Durch die gemeinsame Forschungsgruppe haben wir an BESSY II die Möglichkeit, jederzeit sehr rasch an den viel gebuchten MX-Beamlines Messzeit anzubieten“, sagt Dr. Manfred Weiss, der für die MX-Beamlines an BESSY II verantwortlich ist. Die MX-Beamlines (MX steht für Makromolekulare Kristallographie) sind darauf spezialisiert, winzige kristalline Proben aus organischen, sehr großen Molekülen zu durchleuchten, aus den Daten lassen sich die 3D-Faltungen dieser Makromoleküle entschlüsseln. Tatsächlich zeigt die dreidimensionale Architektur der MHETase einige Besonderheiten: Enzyme wie die MHETase, binden zunächst an ihr Zielmolekül, bevor eine chemische Reaktion eintritt. Für jedes abzubauende Molekül braucht man ein maßgeschneidertes Enzym: „Wir können jetzt genau lokalisieren, an welchen Stellen das MHET-Molekül an die MHETase andockt und wie es dadurch in seine beiden Bestandteile Terephthalsäure und Ethylenglykol gespalten wird“, berichtet Weber.
Nächste Schritte: Aktivität der MHETase steigern
Allerdings sind PETase und MHETase aktuell noch nicht besonders effizient. „Kunststoffe gibt es erst seit wenigen Jahrzehnten in diesem Ausmaß – selbst Bakterien mit ihrer schnellen Generationenfolge und raschen Anpassungsfähigkeit schaffen es nicht in einer so kurzen Zeit, durch den evolutionären Prozess von Versuch und Irrtum eine perfekte Lösung zu entwickeln“, erklärt Weber. „Nachdem wir die Struktur dieses sehr wichtigen Enzyms aufklären konnten, können wir nun auch Varianten planen, herstellen und biochemisch charakterisieren, die deutlich höhere Aktivität als die natürliche MHETase zeigen und sogar gegenüber einem weiteren Zwischenprodukt des PET Abbaus, BHET, aktiv sind", ergänzt Bornscheuer.
Echte Kreislaufwirtschaft
Perspektivisch will Bornscheuer daher daran arbeiten, PETase und MHETase systematisch für ihre Aufgabe, die Zerlegung von PET, zu optimieren. Gert Weber plant, diese Studien durch weitere strukturbiologische Arbeiten zu ergänzen, um die plastikverdauenden Enzyme schrittweise hin zur Anwendung zu bringen. Dazu ist der Zugang zu dem Messstationen und der IT-Infrastruktur des HZB unerlässlich.
Diese Arbeit weist einen Weg hin zu einem „perfekten Recycling“: Künftig könnten solche optimierten Enzyme in biotechnologischen geschlossenen Kreisläufen produziert werden, um PET-Kunststoffe und (perspektivisch) auch weitere Polymere wirklich in ihre Grundbausteine zu zerlegen. Die Kunststoffproduktion wäre dann - Mülltrennung vorausgesetzt - ein geschlossener Kreislauf und nicht mehr vom Rohöl abhängig. Ein Teil des Plastik-Müllproblems wäre damit lösbar.
Publiziert in Nature Communications (2019):"Structure of the plastic-degrading Ideonella sakaiensis MHETase bound to a substrate"; G.J. Palm, L. Reisky, D. Böttcher, H. Müller, E.A.P. Michels, C. Walczak, L. Berndt, M.S. Weiss, U.T. Bornscheuer and G. Weber
DOI: 10.1038/s41467-019-09326-3
Weitere Informationen:
Helmholtz-Zentrum Berlin für Materialien und Energie
Dr. Gert Weber
Forschergruppe Makromolekulare Kristallographie
gert.weber(at)helmholtz-berlin.de
Dr. Manfred Weiss
Forschergruppe Makromolekulare Kristallographie
Tel.: +49 30 8062-13149
manfred.weiss(at)helmholtz-berlin.de
Dr. Antonia Rötger
Wissenschaftskommunikation
Stabsstelle Kommunikation
Tel.: +49 30 8062-43733 / -14759
antonia.roetger(at)helmholtz-berlin.de
Universität Greifswald
Prof. Dr. Uwe Bornscheuer
Institut für Biochemie
Tel.: +49 3834 4204367
uwe.bornscheuer(at)uni-greifswald.de