Coronavirus: BESSY II-Daten beschleunigen die Suche nach Wirkstoffen
Forscher haben die dreidimensionale Architektur der viralen Hauptprotease von SARS-CoV-2 entschlüsselt
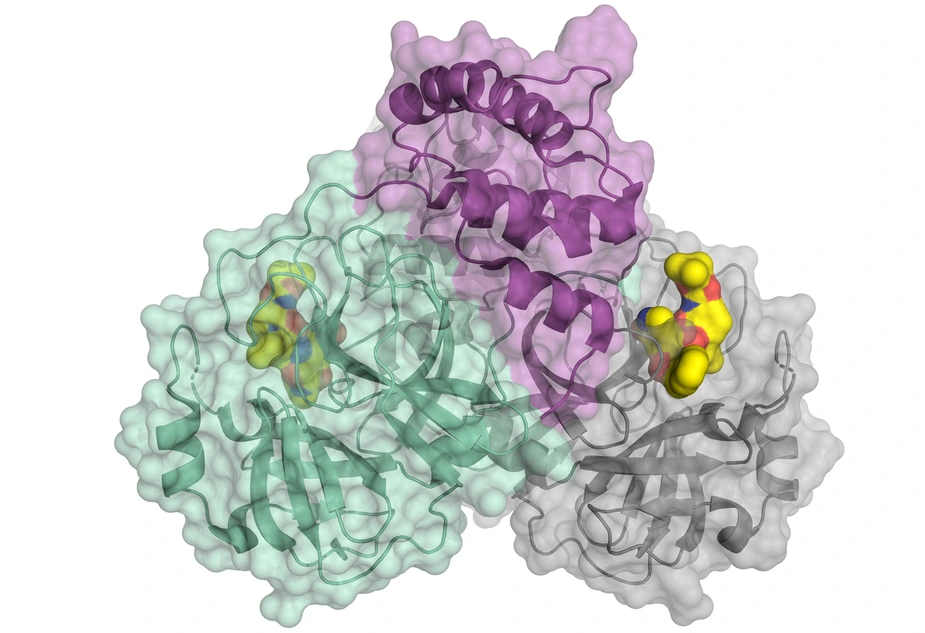
Ein Coronavirus hält die Welt in Atem. SARS-CoV-2 ist hochansteckend und kann schwere Lungenentzündung mit Atemnot (COVID-19) verursachen. Weltweit sucht die medizinische Forschung nach Möglichkeiten, wie man die Vermehrung der Viren mithilfe von Medikamenten verhindern kann. Ein Team der Universität Lübeck hat dafür einen vielversprechenden Ansatz gefunden. Mithilfe des hochintensiven Röntgenlichts der Berliner Synchrotronquelle BESSY II haben sie die dreidimensionale Architektur der viralen Hauptprotease von SARS-CoV-2 entschlüsselt. Dieses Protein spielt bei der Vermehrung des Virus eine zentrale Rolle.
Weltweit forschen Teams daran, Wirkstoffe gegen SARS-CoV-2 zu entwickeln. Wichtig ist dafür die Strukturanalyse von Makromolekülen, die im Virus eine Funktion ausüben. Diese Funktion hängt eng mit der dreidimensionalen Architektur des Makromoleküls zusammen. Kennt man diese dreidimensionale Architektur, kann man gezielt Angriffspunkte für Wirkstoffe identifizieren.
Vermehrung der Viren stoppen
Für die Vermehrung der Viren ist ein spezielles Protein zuständig: die virale Hauptprotease (Mpro oder auch 3CLpro). Nun hat ein Team um Prof. Dr. Rolf Hilgenfeld, Universität Lübeck, die dreidimensionale Architektur der Hauptprotease von SARS-CoV-2 entschlüsselt. Die Forscher haben dafür das hochintensive Röntgenlicht der Anlage BESSY II des Helmholtz-Zentrum Berlin genutzt.
Fast-Track-Zugang an BESSY II
„Speziell für solche hochaktuellen Fragestellungen ermöglichen wir einen „Fast-Track-Zugang zu unseren Instrumenten“, sagt Dr. Manfred Weiss, der die Gruppe makromolekulare Kristallographie am HZB leitet. An den sogenannten MX-Instrumenten, die die Gruppe betreut, können winzigste Proteinkristalle mit hochbrillantem Röntgenlicht durchleuchtet werden. Die Bilder enthalten Informationen zur dreidimensionalen Architektur der Proteinmoleküle. Mit Hilfe von Computerprogrammen lassen sich die komplexe Gestalt des Proteinmoleküls sowie seine Elektronendichte berechnen.
Ergebnisse helfen bei der Wirkstoffentwicklung
Daraus ergeben sich nun konkrete Ansatzpunkte, um Wirkstoffe zu entwickeln. Diese könnten gezielt an Schwachstellen des Makromoleküls andocken und seine Funktion behindern. Rolf Hilgenfeld ist ein weltweit anerkannter Experte auf dem Gebiet der Virologie und hat bereits während der SARS-Pandemie 2002/2003 einen Hemmstoff gegen diese Virensorte entwickelt. 2016 gelang es ihm, ein Enzym des Zikavirus zu entschlüsseln.
Hinweis:
Die Arbeit ist vorab in biorxiv.org (ohne peer review) publiziert, damit die Daten und Ergebnisse allen Forschenden weltweit zur Verfügung stehen. Die Ergebnisse sollen am 20.03.2020 (mit Peer Review) in Science publiziert werden.
"X-ray Structure of Main Protease of the Novel Coronavirus SARS-CoV-2 Enables Design of α-Ketoamide Inhibitors"; Linlin Zhang, Daizong Lin, Xinyuanyuan Sun, Katharina Rox, Rolf Hilgenfeld
DOI: 10.1101/2020.02.17.952879
Weitere Informationen und einen Kurzfilm finden Sie unter
www.helmholtz-berlin.de/pubbin/news_seite
Kontakt:
Helmholtz-Zentrum Berlin für Materialien und Energie
Dr. Manfred Weiss
Forschergruppe Makromolekulare Kristallographie
Leiter des MX-Teams an BESSY II
Tel.: +49 30 8062-13149
E-Mail: manfred.weiss(at)helmholtz-berlin.de
Pressestelle:
Dr. Antonia Rötger
Tel.: +49 30 8062-43733
E-Mail: antonia.roetger(at)helmholtz-berlin.de